Un'altra opzione per un po 'di programmazione è Indigo Toolkit, che ha le librerie Python, Java, .NET e C.
Il seguente esempio di Python prende l'isomero SMILES per Albuterol, che ho ottenuto da PubChem, e rende strutture 2D per tre forme (una con stereochimica non specificata e due stereoisomeri). Le singole strutture sono etichettate con i rispettivi CID PubChem.
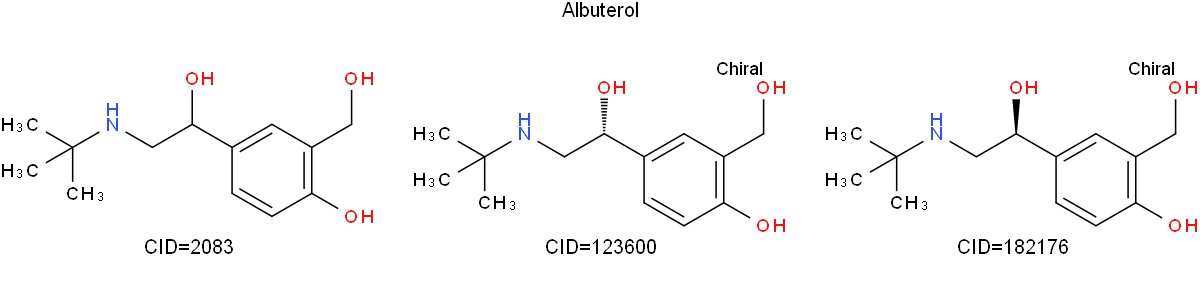
from indigo import * from indigo.renderer import * indigo = Indigo () renderer = IndigoRenderer (indigo) mols = {"2083": "CC (C) (C) NCC (C1 = CC (= C (C = C1) O) CO) O", "123600": "CC (C) (C) NC [C @@ H] (C1 = CC (= C (C = C1) O) CO) O ',' 182176 ':' CC (C) (C) NC [C @ H] (C1 = CC (= C (C = C1 ) O) CO) O '} array = indigo.createArray () per la chiave in mols.keys (): print (key, mols [key]) mol = indigo.loadMolecule (mols [key]) s = "CID =" + key mol.setProperty ("grid-comment", s) array.arrayAdd (mol) indigo.setOption ("render-comment", "Albuterol") indigo.setOption ("render-comment-position", "top") indigo.setOption ("render-grid-margins", "40, 10") indigo.setOption ("render-grid-title-offset", "5") indigo.setOption ("render-grid-title-property", "griglia-commento") indigo.setOption ("render-background-color", 1.0, 1.0, 1.0) indigo.setOption ("render-atom-color-property", "color") indigo.setOption ("render-coloring ", True) indigo.setOption (" render-image-size ", 1200, 300) renderer.renderGridToFile (array, None, 3, "grid.png")